Sunday, November 05, 2006
molecular cloning: banding大量高純度的質體DNA
接下來因應實驗者對質體DNA在數量和純度上的需求,介紹一種稱為”Banding”的質體DNA的抽取方法。所謂的banding是將質體DNA透過高密度介質氯化銫(CsCl)在超高速離心(ultracentrifuge)下形成分離,質體DNA在超高速離心的氯化銫中形成單一個帶狀故名。因為培養細菌的種類和時程會影響最後質體DNA的產量,故以下以一般實驗室常用的菌種XL1-Blue,吃入質體DNA含Amp選質基因為例,根據實驗時間來依序介紹。
(一)第一天下午五點將單珠菌種移植到3 ml的LB培養液(A+T:使用前加入tetracycline:12.5 ug/ml與ampicilin:50 ug/ml),準備或預借之後所需的實驗材料如下:
滅好菌的LB培養液(500 ml/construct)
測OD值所需比色管
Beckman GSA 10 rotor離心用的250 ml離心管( 兩支 /construct :檢查每支管子的O-ring是否完好)
Chloramphenicol(34 mg/ml:in pure ethanol;2.5 ml/construct)
Solution 1(30 ml /construct)
Solution 2(60 ml /construct)
Solution 3(45 ml /construct)
乾淨的紗布數塊
Isopropanolol(0.6倍體積約81 ml /construct)
1xTE(溶解DNA用,pH=8.0、20 ml /construct)
7.5M Ammonium acetate(4 ml /construct)
Beckman SS-34 rotor與其50 ml離心管(兩支 /construct)
100%酒精(30 ml /construct)
RNase A(10 ul /construct)
CsCl(10 g /construct)
Beckman ultracentrifuge用rotor NVT-65、蓋子(cap)、cap封口把手(有磅數指針)、原廠離心管及封口 焊鐵(見附錄一)
1 ml、5 ml注射針數支、18號、21號針頭、一般15 ml離心管
EtBr(1 ml /construct)
手提式紫外光燈
l-butanol(pure & saturated:含水)
透析用DNA袋子、夾子和裝4公升TE溶液的容器,stir bar等....
(二)第二天上午11點將3 ml的菌液取1 ml加到500 ml的 LB培養液(A+T),再放回培養箱進行培養。9~10小時之後進行菌液OD值比色,以乾淨的LB( A+T)為基準做校正,確定細菌生長週期達到plateau(OD值約0.8~1.0之間)。此時加入2.5 ml的 Chloramphenicol(final concentration=170 ug/ml), 再放回培養箱養16小時 。Chloramphenicol的作用在於使細菌停止細胞複製,儘量製造更多的質體DNA,。
(三)第三天下午2點回收細菌,將菌液分裝到250 ml的離心管內,進行4500 rpm(Beckman GSA 10 rotor)、4度C離心20分鐘。倒掉上清液留下下方的pellet,敲打試管或vortex使pellet完全散開,再依序加入S1 30 ml,invert數次,靜置室溫15分鐘;S2 60 ml, gently invert數次,靜置冰上15分鐘;S3 45 ml, gently invert數次, 靜置冰上15分鐘。之後進行5000 rpm(GSA 10 rotor)、4度C離心20分鐘。
(四) 以雙層紗布過濾上清液到另一個乾淨的250 ml離心管,如過濾不乾淨可考慮重覆離心、紗布過濾兩次,確定過濾後的液體澄清,沒有白色沈積物。再加入0.6倍體積的 Isopropanolol(約81 ml), invert數次,靜置冰上15分鐘。之後4度C離心5000 rpm(GSA 10 rotor)20分鐘,倒掉上清液,這時可見瓶底有透明pellet,倒扣瓶子Dry乾液體數分鐘,加入10 ml TE搖晃均勻,最後將10 ml液體(增加回收率可以分4、3、3 ml三次)放到新的50 ml離心管(for SS-34 rotor)。
(五) 加入4 ml 的7.5M Ammonium acetate, 搖晃均勻後靜置冰上10分鐘,4度C離心4500 rpm(SS-34 rotor)10分鐘,此時下方pellet為殘留的細菌染色體與蛋白質,取上清液到新的50 ml離心管(for SS-34 rotor),加入30 ml 100%的酒精,放置負20度C冰箱至少兩小時;再離心 4度C離心10000 rpm(SS-34 rotor)40分鐘;到掉上清液,真空45度C dry乾1小時(可使用保鮮膜封口再行戳洞),之後加蓋保存負20度C冰箱。
(六)解凍後加入10 ml TE溶解下方pellet(增加回收率可以分4、3、3 ml三次),將溶好DNA的TE移到新的15 ml離心管(先取出5 ul當控制組),加入10 ul的RNase A,37度C作用1小時,取5 ul run gel(這步驟可以檢視RNase A是否將RNA清除乾淨)。
(七)精秤CsCl 10克(誤差再0.001g之內)放入50 ml的離心管,將10 ml的TE加入,搖晃至粉末全部溶解;再用5 ml針筒將液體全部加入 Beckman ultracentrifuge 專用離心管( NVT-65 rotor專用),加入時液體由管壁緩慢留下,眼睛檢視絕對不可以有氣泡產生。之後用1 ml針筒將EtBr(有毒物質請小心操作!)加入約0.7~1.0 ml,因為上超高速離心一定要兩邊試管重量相等,所以後果不堪設想!所以加入EtBr的質量提供平衡兩試管重量的好機會(平衡的兩試管誤差應小於0.01克),方可上超高速離心機。
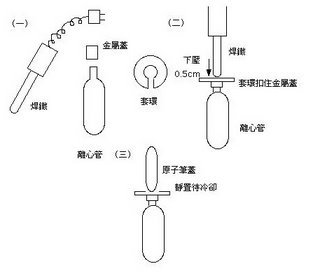
(八)小心用Beckman原廠焊鐵封口(見左圖),封口後可以用手用力壓壓看測試是否完整。封口後invert數下,使EtBr混合均勻,包覆錫泊紙以阻絕光線。上機前在確定兩試管重量相等,方可進行超高速離心。 NVT-65 的 rotor 每支離心管都要加 Cap(with O-ring),用原廠把手鎖緊,確定扭力磅秤讀數約120磅,上機進行超高速離心,離心參數為 20度C、65000 rpm 6小時。
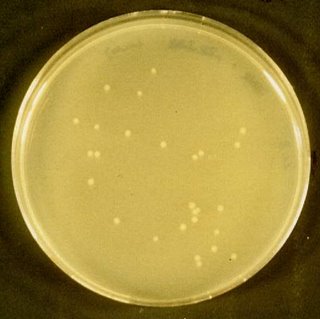
(九)解開 Cap,小心翼翼(勿晃動)將離心管用夾子抽出,鋪好bench紙,打開紫外光燈,此時可見三條主要亮帶(見下圖)先用21號針頭由離心管上方插入(release pressure),在用18號針頭、5 ml針筒由質體DNA的band下方刺入離心管,抽出橘紅色液體約1~2 ml(吸取時可旋轉針頭),放到新的15 ml離心管。

(十)用dropper取兩倍體積(約4 ml)的 Saturated l-butanol(含水),invert混合均勻後室溫離心2000 rpm、3分鐘,用dropper吸掉上清液;再用dropper取兩倍體積(約4 ml)的 Pure l-butanol(不含水), invert混合均勻後室溫離心2000 rpm、3分鐘,用dropper吸掉上清液,這步驟可以去掉大部分的EtBr,準備乾淨的dialysis bag,將質體DNA用p1000移進袋子裡,放到4公升的TE中進行透析,此過程在4度C冰箱中進行,將裝好TE的桶子放在電磁攪拌器上,下方stir bar維持旋轉,6~8小時更換一次乾淨的TE,如此重複3~4次。
Saturday, November 04, 2006
molecular cloning 如何不用kit抽取質體DNA?
這裡要介紹的不是教你如何使用kit,因為鸚鵡學禪的方法在坊間所有販售的kit裡已經有清楚的說明書。如何不用column也可以抽出質體DNA?基因工程實驗常需要挑很多的「克隆(clone)」,挑選的方式不外PCR或colony hybridization這兩種,當實驗者挑到很多可能的「克隆」時,就必須將這些「克隆」進行質體DNA的抽取,以便之後的檢定工作。這裡介紹一個簡便又經濟的抽取方法(特別感謝榮總腎臟科 林堯彬醫師提供)。使用步驟如下:
(1) 將單一菌落(single colony)轉植到1 ml的 LB(事先一實驗設計需要加入特定的抗生素如ampicillin、tetracycline),放在37度C、240 rpm培養16小時。(可以用一般1.5 ml的eppendorf,用小釘子將蓋子口打個小洞)。16小時候將整支試管拿去離心一分鐘(16,000 g)。倒掉上清液,再用震盪器將底部的pellet打散(這步驟很重要,一定要確定打散喔!)。
(2) 加入 100 ul 的溶液S1,invert數下,使之混合均勻。(配方見下附註)。
(3) 加入 200 ul, 的溶液S2,小心輕輕地 invert數下(因為此時細菌的細胞壁被NaOH打破,細菌的染色體DNA外露,為避免斷裂,千萬不可太用力。),靜置室溫5分鐘 。
(4) 加入 200 ul, 的溶液S3,小心輕輕地invert數下後靜置冰上5分鐘。此時細菌的蛋白質和高濃度的醋酸鉀形成白色沈澱。(因為細菌的染色體DNA很長且包附在蛋白質上,會被一起抓下來)。
(5) 4oC離心16,000g 、 5 分鐘,將白色沈澱與上方質體DNA分開。
(6) 準備新的離心管(eppendorf),每隻試管加入1 ml的 酒精(純度至少95%)。
(7) 將步驟(5)的上清液移到酒精中,靜置冰上約30分鐘。
(8) 4oC離心16,000g 、 5 分鐘,倒掉上清液,留下下方pellet。
(9) 用70% 酒精 500 ul清洗pellet。
(10) 4oC離心16,000g 、 5 分鐘,倒掉上清液,用p200儘可能吸乾所有酒精液體。
(11) 倒扣試管室溫下靜置30分鐘,使底部pellet儘量乾燥。之後加入20~50 ul 的TE buffer(pH=8.0)。
理論上,此法所得到的質體DNA,其純度已經可以進行DNA限制酵素(restriction enzyme)的切斷實驗。因為細菌的染色體和蛋白質分離只依賴離心作用,所以質體DNA的純度並不如一般市面上販售的column kit,此方法只建議使用在初步篩選所有可能的「克隆」。如果實驗者挑選到正確的「克隆」,就質體DNA的純度和保存觀點,還是建議將正確「克隆」用column抽取。
附註:
Solution 1(500 ml):秤重Glucose 4.5(g)、加入450 ml純水 stir至完全溶解;在加入0.5M EDTA (pH=8.0)12.5 ml、 1M Tris-HCl (pH=8.0)10 ml,最後補水到500 ml(autoclave後再補滅菌水),使用前加入RNAse(final....100U/ml)4oC保存。
Solution 2(1000 ml):配置0.4N NaOH 500 ml,2% SDS 500 ml(使用無菌純水)。等體積混合兩溶液即可。
Solution 3(200 ml):秤重potassium acetate 58.89(g),加入無菌純水120 ml,再用醋酸glacial acetic acid調整pH值為5.0(約23 ml),最後補無菌純水到200 ml,即為0.5M potassium acetate buffer。